Les images Sentinel 2 sont composées de différentes longueurs d’onde permettant l’analyse de la végétation au sol

Pour estimer les variables bio-physiques dignes d’intérêt on utilise les bandes du domaine visible, red-edge, proche infra-rouge et infra-rouge, or la résolution de ces bandes n’est pas la même, certaines ont une résolution de 10 et d’autres une résolution de 20m.
Or pour travailler ces images, on doit nécessairement avoir des bandes à la même résolution.
Une méthode simple consiste à transformer une bande de 20m en une bande de 10m en procédant à une interpolation bilinéaire des pixels de 20m après avoir divisé leur taille par 4 (Dans un pixel de 20 m il y a 4 pixels de 10m de coté)
L’interpolation consiste à attribuer à un pixel la valeur d’un pixel la moyenne des x pixels voisins, si x=3 alors on utilise une fenêtre mobile de 9 pixels de coté que l’on déplace sur toute l’image pour calculer la valeur de chaque pixel. Ca s’appelle un filtre convolutif. Il permet de lisser une image en supprimant les éventuels pixels bruités, l’inconvénient est qu’il entraîne un effet de flou sur l’image.
Voyons ce que cela donne sur une image exemple:
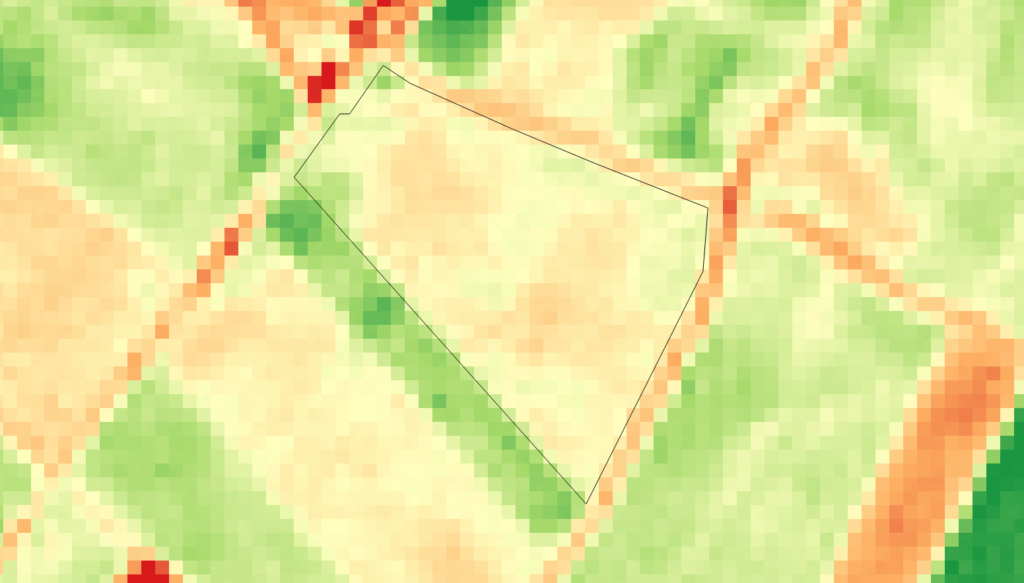
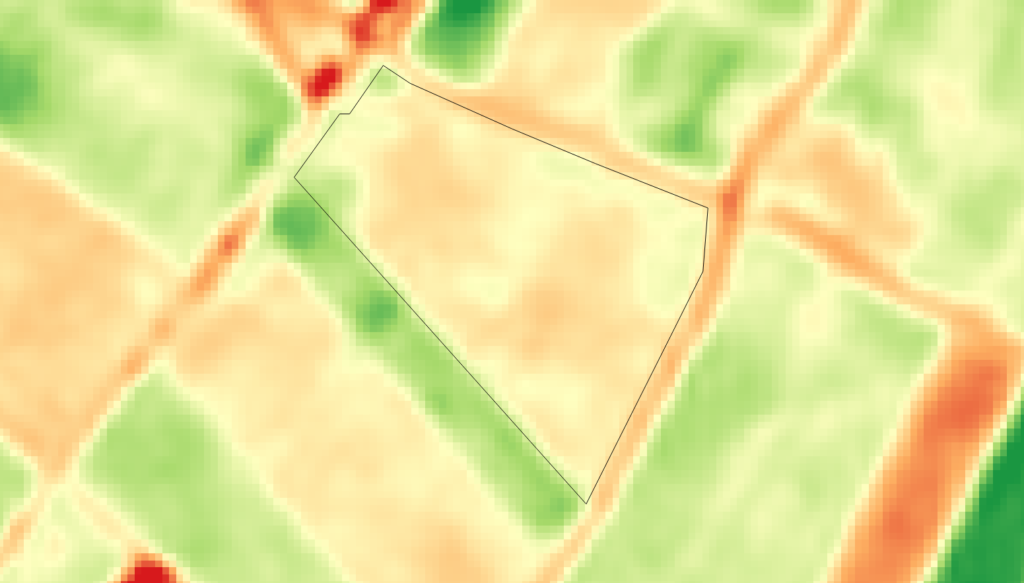
Le rendu est plus lisse, mais plus flou, en augmentant la valeur de x du filtre, on augmente encore ce niveau de flou, mais on supprime le bruit.
L’avantage de ces filtres est qu’ils sont très faciles à mettre en œuvre et le calcul est très rapide sur une image de 120 Millions de pixels.
Afin d’exploiter la résolution des bandes de 10m sans perdre l’information spectrale des bandes de 20m, on utilise différentes méthodes de pansharpening ou en français fusion des bandes. Il en existe à peu près autant que de scientifiques qui les étudient. Il faut alors procéder à des calculs matriciels parfois très complexes et lourds qui, pour une image Sentinel peuvent prendre des heures, ce qui est inconcevable dans une exploitation de masse des images. Il faut donc trouver un compromis entre précision des résultats et temps de calcul.
Après avoir testé de nombreuses méthodes (IHS, Broovey, ATPRK, PCA, GSA, Support Vector Regression, Artificial Neural Networks), je n’ai pas échappé à la règle, j’ai mis au point une méthode “hybride” combinant une régression non linéaire des bandes 20m à partir des bandes 10m, puis j’y ai ajouté la matrice des résidus transformée en 10m à l’aide d’un filtre convolutif dit “cubic”.
On obtient alors l’image ci dessous:
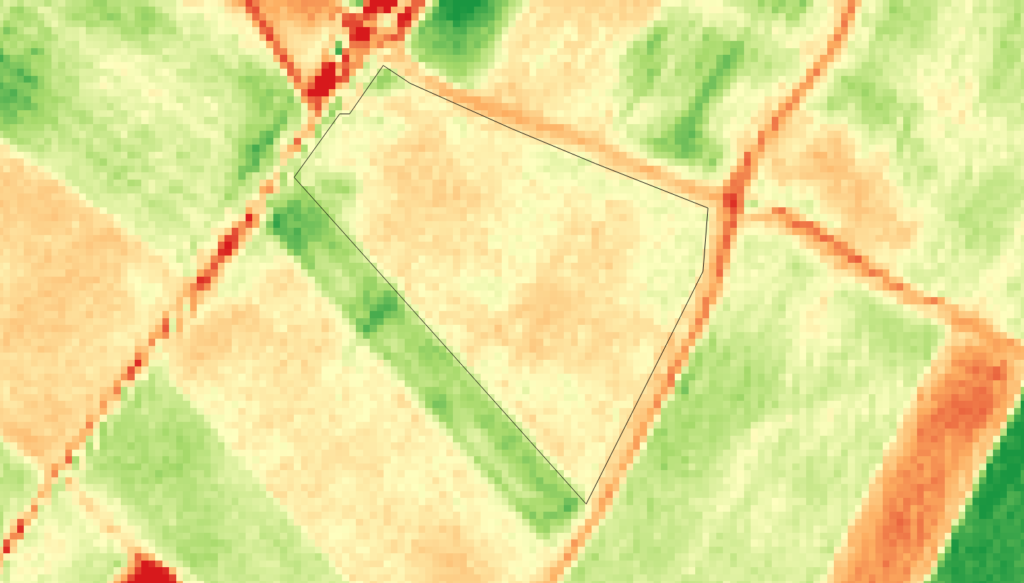
On constate que on retrouve bien la résolution à 10 m sans l’effet de flou du filtre. Cette résolution est obtenue grâces aux bandes 10 m utilisées dans le calcul. Reste néanmoins à vérifier si l’information spectrale de la bande en 20m est toujours là, on peut utiliser des indicateurs spécifiques (Spectral Angle Mapper, ERGAS, PSNR, UQI), je n’y ai pas trouvé un intérêt particulier et je préfère rester sur le bon vieux RMSE (Root Mean Square Error) et le coefficient de corrélation (R2)
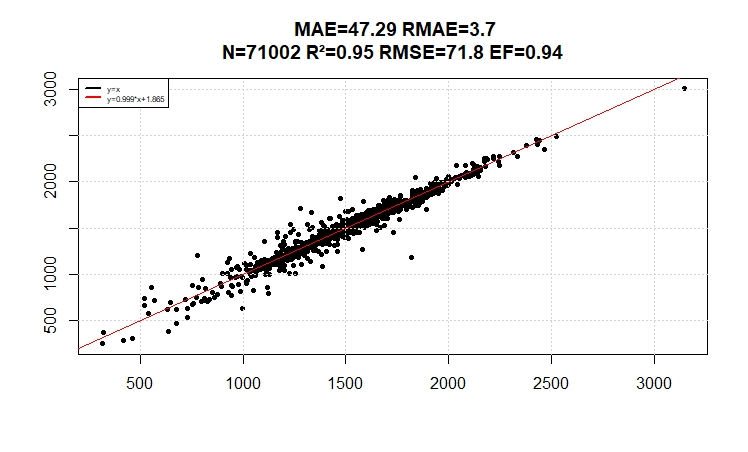
Les réflectances varient de 0 à 10000 dans les images Sentinel, sur ce graphe l’on peut constater la bonne corrélation et l’efficience du modèle de Pansharpening.
En comparaison ci dessous, la corrélation entre la bande en 20m et la bande interpolée avec le filtre bilinéaire (flou)
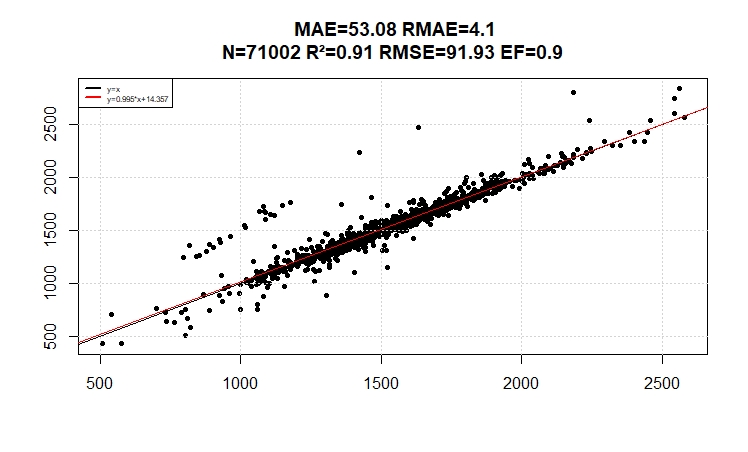
Le résultat, même s’il est moins bon que la méthode de pansharpening n’est pas si mauvais, mais c’est l’effet visuel qui n’est pas appréciable et qui se voit sur les rendus aux utilisateurs, à savoir, soit sur des pixels de 20m l’effet de pixellisation vu sur la le 1er graphique, soit l’effet de flou vu sur le 2e.
En conclusion, cette méthodologie permet de produire des cartes de variables biophysiques (Indice foliaires, biomasses, modulation de fertilisation) plus précises et plus agréables à regarder et donc sans doute plus compréhensibles au premier coup d’œil pour l’utilisateur.

